水通道蛋白库
专为发现新型水通道调节剂而设计
1 348 种化合物
该化合物库旨在寻找针对水通道蛋白(Aquaporins)AQ1、AQ4、AQ5 单体的末端链和 AQ5 的内部中央孔的分子,这些分子具有与 AQ1 和 AQ4 中的分子相似的物理化学性质。由于已知的针对这类靶点的有效抑制剂较少,无法建立定性的 QSAR 模型或药效团模型。我们采用高通量筛选对接方法来寻找新的潜在抑制剂,进行 2D 相似性搜索以富集与报道的活性物质类似的化合物。
库的设计
对接优化与计算
在分析了 11 种可用的 X 射线结构(1H6I、1IH5、2ZZ9、3GD8、4NEF、3D9S、5DYE 等)后,选择了三种蛋白结构(3D9S、1IH5、3GD8)。然而,众所周知,开放/关闭状态的转变对某些突变非常敏感。这些数据描述了位于通道内部的几个热点,这些热点被用于在蛋白质结构完善的初级阶段准备对接模型,当然前提是这些残基参与了相互作用,而非仅仅增加螺旋的刚性。此外,利用 FT-MAP 服务器对每个单体的整个表面进行了探测,这是一种非常准确和值得信赖的结合位点识别工具。它基于对小型有机探针(溶剂、饱和和不饱和循环等)与目标表面之间的能量相互作用的评估。
水通道蛋白库的设计采用了两种不同的方法:灵活对接和高通量筛选。灵活对接协议假设位于 AQ5/AQ4/AQ1 蛋白质通道入口处的侧链部分具有自由的可旋转运动。灵活性区域覆盖了四聚体结构中通道中心周围约 5Å2 的范围。根据残基突变研究和 FT-MAP 计算,选择了一个网格框(测定结合区域)的中心。为了扩大可能的结合位点的体积,我们将一组在文献和 ChemblDB 中公开的真正抑制剂与已知目标相互作用的化合物一起进行对接(AqB013、Bumetanide, Zonisamide, Arbidol 和 Acetazolamide 取自文献,一些神经递质,如多巴胺、肾上腺素和血清素,根据我们与学者的合作选择)。
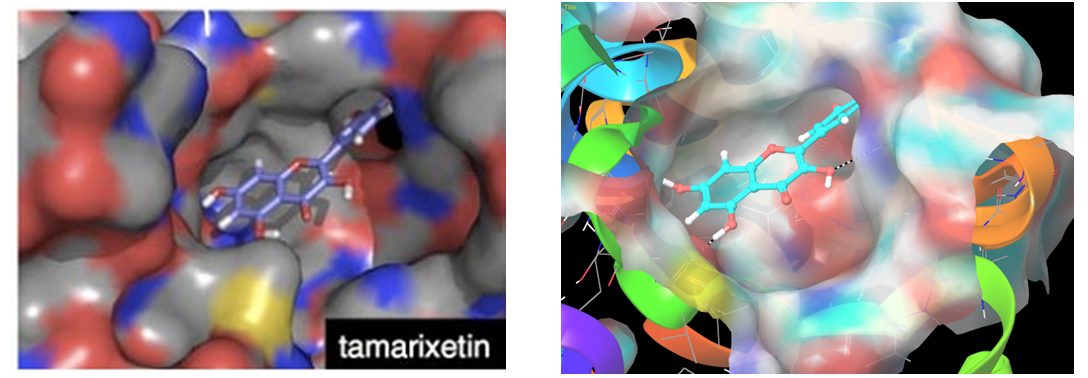
已报道的 tamarixetin 结合模式(左)和对接计算后获得的 hit 分子实列(右)。
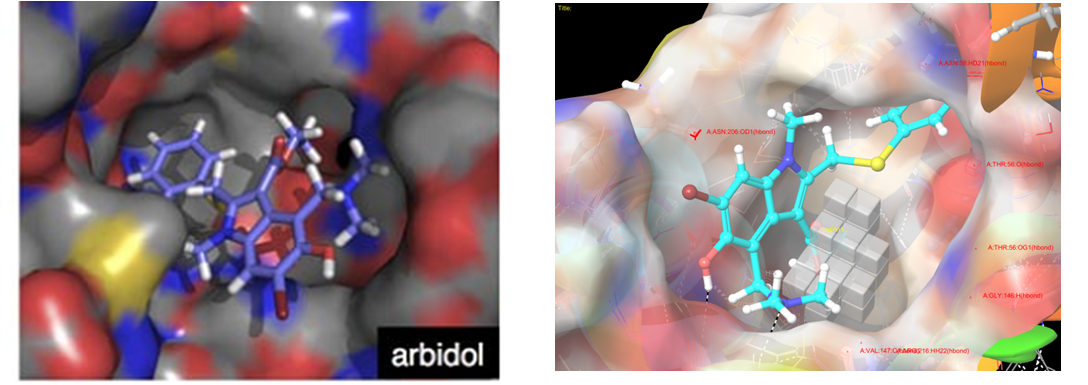
一些负责底物/抑制剂结合的残基 - G146、V147、R216、T56(Asn58、Gly209、Val141)对 arbidol 的结合也很重要。
通过这种方式,我们实现了通道闸口在结合位点处更为适宜的构象,随后将这些优化后的结构应用于筛选程序中,此时采用的构象评估参数和生成构象数量稍有简化。以下简要介绍 AQ5 模型对接过程中的参数设置示例。在构成过滤筛网的约束条件中,值得一提的是通道内部的疏水核心与一组特定氨基酸的部分匹配(对于 AQ5 的 site 2 位点:ARG:188、ASN:120、GLY:181、HIS:173、VAL:119; 对于 AQ5 的 site1 位点:GLN:81、HIE:67、PRO:62、SER:152、THR:150、THR:150、TYR:90、TYR:90)。所有这些细节在图表中均有展示。那些携带疏水片段并满足另一条件(即与上述列表任一残基形成 2-3 个氢键相互作用)的化合物,将晋级至下一阶段更为精准但耗时的对接程序。化合物的评分是经验值、能量后计算、空间效应及氢键作用力后处理结果的综合提现。评分越高,表明配体与靶标结合的可能性越大。
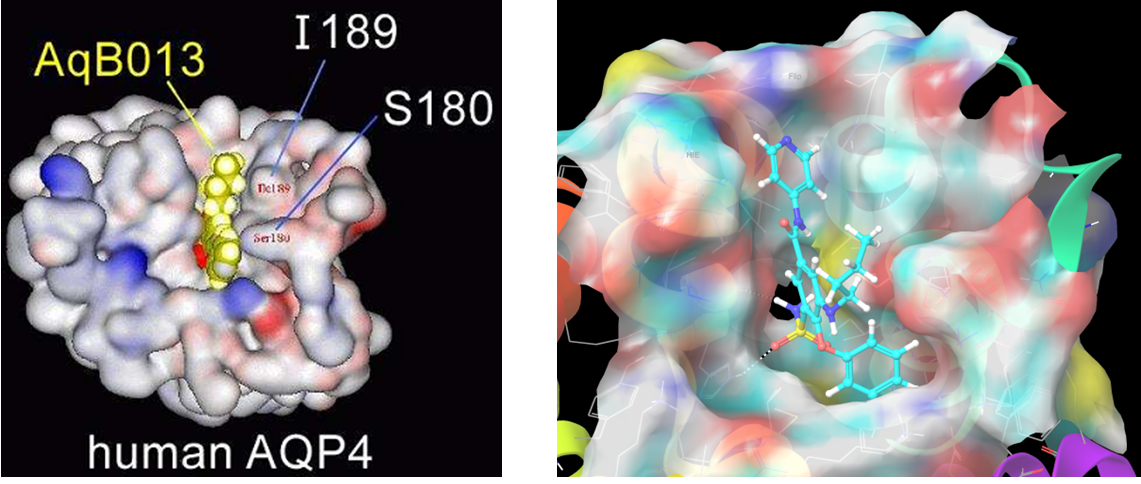
化合物 AQB013(与布美他尼相同)与 AQ4 的第二个位点对接。更理想的残基组合是 I189、S180、Phe175 和 Arg261,它们形成了 AQ4 通道的对立门。



